Analyses de l’ARN/ADN pour identifier ou confirmer des biomarqueurs et des critères d’évaluations cliniques
Afin de prescrire le traitement le plus adapté en matière de cancer, un ensemble de diagnostics sont réalisés pour déterminer la présence de la pathologie, sa nature, sa forme ou sa classification. Ainsi, des thérapies spécifiques sont dédiées à des sous-classes particulières de cancers. Cette approche de médecine de précision existe principalement pour traiter les cancers du sein, et elle a permis une amélioration significative du taux de survie à 10 ans pour ce cancer : de 40% en 1971 à plus de 80% en 2021.
Néanmoins, la résistance ou l’insensibilité au traitement reste un problème important. Cela est dû à l’absence de marqueurs prédictifs de la résistance au traitement ou au manque de biomarqueurs associés au suivi des patients pendant le traitement.
OncoSNIPE® un programme de recherche collaborative sur l’identification de biomarqueurs en oncologie
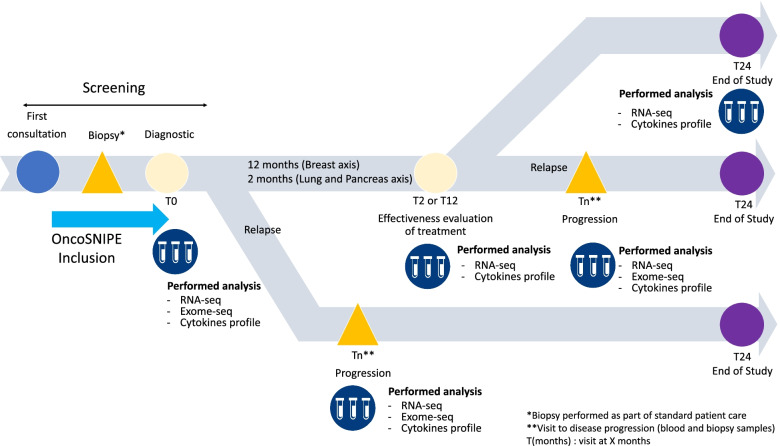
Dans le cadre du programme de recherche collaborative OncoSNIPE®, certains critères d’évaluation clinique ont été associés à l’échantillonnage (initial, suivi, progression de la maladie) et, après certaines analyses, à des biomarqueurs spécifiques. Les analyses seront effectuées dans chaque cohorte (cancer) indépendamment. L’objectif est d’identifier des biomarqueurs précoces (initiaux, prédictifs) ou tardifs (suivi, surveillance) associés à la résistance au traitement pour chaque critère d’évaluation clinique.
La résistance est définie comme une non-réponse tumorale selon les critères RECIST (Response Evaluation Criteria In Solid Tumor) ou l’absence de réponse pathologique complète (pCR).
Ce protocole d’étude a conduit au développement d’un pipeline optimisé et standardisé d‘outils bioinformatiques et biostatistiques pour effectuer des analyses de routine de l’ADN et de l’ARN des données de séquençage.
L’objectif des analyses exome/ADN est l’identification de mutations putatives de l’ADN, comme les InDels.
Dans le cas des analyses RNA-Seq, différentes étapes (normalisation, cartographie, comptage) sont nécessaires avant l’analyse des gènes différentiellement exprimés, ou l’identification des modifications de l’ARN comme l’épissage ou l’édition de l’ARN. Ces analyses conduiront à l’identification ou à la sélection de marqueurs ARN putatifs selon des critères et des objectifs prédéterminés : clustering, ou recherche exploratoire pour mettre en évidence des voies métaboliques ou de signalisation déjà connues.
L’ensemble du protocole d’étude appliqué dans ce programme de recherche collaborative a été détaillé dans une publication récente : “OncoSNIPE® Study Protocol, a study of molecular profiles associated with development of resistance in solid cancer patients” (Vachenc et al, BMC Cancer 2022).
Acobiom utilise son expertise pour développer des thérapies ciblées

Dans le cadre d’un projet collaboratif comme OncoSNIPE®, ou dans le cadre d’un essai clinique évaluant l’efficacité d’une nouvelle thérapie, ACOBIOM est convaincu que la recherche et l’identification de biomarqueurs omiques dans le sang périphérique est une stratégie pertinente pour identifier et sélectionner une sous-population de patients, répondeurs à une thérapie spécifique dans une optique d’efficacité à long terme.
Ainsi, l’identification de biomarqueurs prédictifs permettra d’améliorer les résultats des traitements actuels, de réduire leurs effets secondaires, et d’identifier le développement de résistances.
Cette approche qui peut suivre des critères d’évaluations cliniques pourrait également permettre l’identification de biomarqueurs pronostiques associés à de nouvelles thérapies.
En effet, ACOBIOM peut exploiter les échantillons sanguins et les données associées, collectés lors des essais cliniques, pour optimiser le développement de thérapies ciblées ou d’immunothérapies dans des sous-groupes de patients prédéfinis et ainsi, dé-risquer les nouveaux projets et maximiser les chances de succès des essais cliniques.
Dans ce cas, la société utilise son approche propriétaire d’apprentissage machine basée sur les données omiques pour déterminer les différentes sous-populations de patients, pour identifier les « bons » et les « mauvais » répondeurs et pour établir un score d’une signature génique sanguine afin de pronostiquer les réponses des patients et d’ouvrir la voie à la personnalisation du traitement.
Cette stratégie d’enrichissement prédictif réduit la variabilité de la population de patients étudiée et permet de sélectionner les patients qui sont les plus susceptibles de répondre à un médicament spécifique par rapport aux autres patients atteints de la pathologie traitée.

